SOLUTION
検査室支援情報
検査の樹 -復習から明日の芽を-
18.「悪液質」思いちがいも、本態疾患の治療に抵抗を示す
Vol.18-2「悪液質」診断への探索
第1部Vol.18-1「悪液質」の実状ではCACの実状を中心に述べてきたが、第2部では悪液質発症初期の徴候をとらえる分子探索に焦点を当てたい。
6.CACのバイオマーカー
CACのバイオマーカー:満たされていない医療ニーズ
CAC分野における初期バイオマーカーの同定は、がん診療への貢献性が極めて高く、かつ不可避事項でもある。このため、多くの研究者が多方面からの探索に鎬を削っているが達成は困難を極めている。
分子バイオマーカーとしては、炎症メディエーター、循環因子、代謝物、タンパク質、アミノ酸、脂質、マイクロRNAなど数多くの分子が標的となり、血液、唾液、尿、便および組織などから分析されている。しかし、CACは多因子が多様に交錯し複雑な代謝反応を示すため、統括因子を特定することは困難な実状が垣間見える。目的達成には、何らかのグループに分け複数のバイオマーカーを設定する必要があるのかもしれない。炎症メディエーターとしては、IL-6, TNF-α、IL-1β、CRP,アルブミンなどがあげられるが 炎症メディエーターは腫瘍細胞と宿主細胞との双方から放出され、CACでは頻繁に上昇するため難攻している[105] [106]。また、血清CRP値の上昇とアルブミン濃度の低下で構成するグラスゴー予後スコアなど、複数因子からスコアを算出する研究も試みられている[107][108]。免疫系では、がんの予後指標である好中球対リンパ球の比率(NLR)を進行性結腸がん、肺がんおよび前立腺がん患者での体重減少と悪液質および血清中の25-ヒドロキシビタミンD(25(OH)D)濃度との関連性などがバイオマーカーとして提案されている[109]。
炎症因子の他に循環タンパク質として、CACの発症との関係より、ZAG(:亜鉛-α2-糖たんぱく質zinc-α2-glycoprotein)なども調査されている。ZAGは、WATの褐変と脂肪分解を促進する脂質動員因子である[110][111]。ヒトのがん性悪液質では循環アクチビンA(ActA)が高値を示す。また、ActAレベルは、がん患者の体重減少と骨格筋密度と相関性が見られる[112]。さらに、ミオスタチン[113]、GDF15(成長/分化因子15Growth/differentiation factor 15)は骨格筋萎縮(アクチビンA、およびミオスタチン)、食欲不振、脂肪分解(GDF15)に関与する非定型トランスフォーミング成長因子ベータ(TGF-β)スーパーファミリーのメンバーでもある[114]。成長分化因子 15 (GDF15)は、ストレス関連ホルモンとして浮上している。炎症性疾患、心血管疾患、およびがんにおいて、GDF15 は疾患の結果のバイオマーカーである。GDF15は、エネルギー恒常性、体重調節にも関与しており、悪液質において独特の役割を果たしている。また、その受容体であるグリア細胞株由来神経栄養因子(GDNF)ファミリー受容体α様(GFRAL)が最近発見され、その代謝機能が明らかになってきた。
PTHrP(副甲状腺ホルモン関連ペプチド)はWATの褐変を誘発し、アンギオテンシンⅡは筋肉の消耗を誘導する、LPS結合タンパク質は腸管バリア機能不全のマーカーとして、悪液質の発症と患者の生存率について興味深い予後値を示した[115][116]。リポカイン2は食物摂取を調節し、膵臓がん患者の好中球の増加、筋肉の消耗、生存と相関する[117]。INSL3(:インスリン様3 Insulin-like 3)は食欲不振を促進し、血漿レベルは膵臓がん患者の食欲不振の重症度と相関する[118][119]。
また、腫瘍が産生するPIF(:タンパク質分解誘導因子proteolysis-inducing factor)[55]やLMF(:脂質動員因子lipid mobilizing factor)[120]がある。
Bindelsらは、化学療法とは無関係に腸機能(腸管透過性、上皮細胞のターンオーバー、腸管免疫、微生物叢異常)に大きな変化が生じる、これらの変化は食欲不振とは無関係に発生し、インターロイキン6によって引き起こされる。腸機能障害は、抗炎症細菌(Faecalibacterium prausnitzii)または腸管細胞の再生に関与する腸管ペプチド(テデュグルチド、グルカゴン様ペプチド2類似体)による治療に抵抗性である。悪液質マウスおよびがん患者で上昇しただけでなく、血清IL-6レベルと強い相関関係があり、これらの患者の死亡および悪液質の発生を予測する。全体として、がん悪液質で腸管恒常性の重大な変化が発生している。これは人間にも関連している可能性がある。さらに、腸内細菌叢異常に関連するがん悪液質の新しいバイオマーカーとしてリポ多糖結合タンパク質を指摘した[116]。
悪液質は筋肉および脂肪組織の消耗に関係するため、アミノ酸、コラーゲンおよびタイチン(チチンまたはコネクチン)断片、カルノシンジペプチダーゼ1、グリセロール、遊離脂肪酸など、これらの異化プロセスに由来する代謝産物が上昇する[115][120]。
スフィンゴ脂質[121]やリポタンパク質[122]など、他の代謝産物が最近特定されている。スフィンゴ脂質、リポタンパク質、アミノ酸はいずれも悪液質発症の初期マーカーと考えられ重要性が提起されている。さらに、近年注目を集めている、マイクロRNAおよびその他のncRNAが新規マーカーとして浮上している[122][123][124]。
ここに引用したバイオマーカーの中で近年注目を集めているのは、GDF15とmiRNAのグループである。しかし、個々のがん種での反応性、腫瘍発症からの時間的経過、個々の患者における腫瘍発症時の生理状態の差異など多くの課題も残る。
その中でも、IL-6、TNF-α、IL-1β、CRPおよびアルブミンが最も研究されているが、それらとがん性悪液質との相関関係はヒトでは明確化されているとはいえない[105]。
免疫系に関連するその他の因子もバイオマーカーとして提案されており、例えばがんの予後指標である好中球対リンパ球比は、進行した大腸がん、肺がん、前立腺がん患者を対象とした後ろ向き研究で、最近体重減少や悪液質との関連が示された[109]。
Millerらはメタボロミクス手法を追求し、体重が安定している患者と比較して、体重減少患者では中鎖リゾホスファチジルコリン(LPC)グリセロリン脂質などの血漿リゾ脂質の増加を報告した[125]。同様に、Calaらは、短鎖LPCの有意な減少とアミノ酸、特にヒスチジン、リジン、トリプトファン誘導体の減少を特定し、悪液質との関連性を報告した[126]。さらに、さまざまな画像診断法から得られる定量的な体組成データと組み合わせると、マルチオミクスの応用が特に強力になると考えられる。
新しいオミクス研究により、がん性悪液質に関する知識が深まり、早期診断のための潜在的なバイオマーカーのリストが増えている。一般的な悪液質とがん特有の悪液質の両方の誘発因子に焦点を当てることで、これらのアプローチにより、がんの種類に応じた新しい標的療法の開発が可能となる。
より具体的には、メタボロミクス研究[127]により、すでに述べたマイクロRNA[123]、スフィンゴ脂質[121]、リポタンパク質およびアミノ酸の変化[122]などの悪液質の非侵襲的マーカーを特定することが可能となり、目的達成も夢ではないかもしれない。
さらに、悪液質の診断にはさまざまなバイオマーカーと臨床徴候の組み合わせが必要になるだけでなく、前臨床モデルで強調されているように、腫瘍の種類によってマーカーが変化する可能性がある。例えば、食欲不振はC26(Mice bearing the Colon-26)保有動物にはみられ、LLC(Lewis lung carcinoma : RCB0558)保有動物にはみられないが、どちらの腫瘍も悪液質を引き起こす。
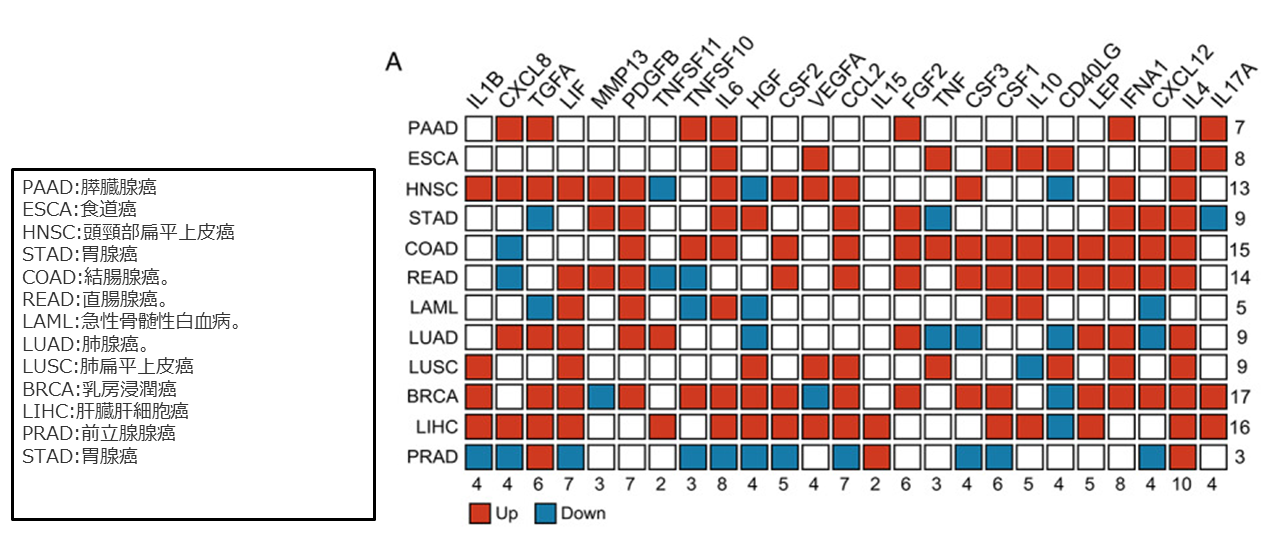
図8 The Cancer Genome Atlas(TCGA)の12種類の腫瘍における25個のCIFの発現パターンを要約した概略図
CIF:cachexia-inducing factor
J Cachexia Sarcopenia Muscle. 2020 Aug;11(4):947-961 より和訳引用.
図8に12種類の腫瘍における25個のCIF(Cachexia-Inducing Factor)の発現パターンを要約した概略図を示したが、腫瘍種によって幾つかの特徴的発現パターンの差異が認められる。したがって、これはメカニズムが複数あるということを示しており、すべてのがん患者に有用な1つの理想的なバイオマーカーを特定することは極めて困難と思われる。引用されたバイオマーカーの中で、現在大きく注目を集めているのはGDF15であり、その診断価値に加えて、治療ターゲットとして積極的に研究されている[128][129]。したがって、まだ多くの作業が必要であり、明確な時系列が明らかにされない限り、がん患者とその管理のパラダイムを変えることができるバイオマーカーを特定することは非常に困難である。
バイオマーカーの探索には人間の患者に近い動物モデルを開発する手段もあるが、悪液質は複数の組織の全体的な混乱であり、多くの場合、複数の要因(インスリン、レプチン、グレリンなど)に対する全身的な抵抗性を引き起こす。複数の回路が故障すると、患者は帰還不能点に近づき、健康が確実に失われ、治療介入はおそらく不可能であるように見える[130]。
7.がんにおける遺伝子変異
癌(がん)は、これまで多数の報告が見られるように、患者の多くに遺伝子変異が認められている。がんは、この遺伝子変異した遺伝子から翻訳されたタンパク質により形成される。がん細胞は異常に増殖するだけでなく、抗アポトーシス性あるいはアポトーシス促進性の遺伝子またはタンパク質の発現を抑制するなど、正常な代謝とは異なるがん細胞に好都合な代謝経路が構築される。このように、がん細胞には既に正常な代謝経路とは異なる、シグナル伝達、代謝調節、代謝経路が存在する。これらの大きな基点となるシグナル伝達タンパク質Rasや、損傷した遺伝子のゲートキーパーとして働き、悪性細胞のアポトーシスを開始する調節遺伝子p53などの主要な遺伝子にも変異が見られる。このため正常な機能、代謝、調節が出来なくなる。
この他にも変異が見られる遺伝子は多く、主な遺伝子としては、BRAF, RAS, PTEN, PI3KCA, TP53, RET, CHEK2, CDH1, NBS1, RAD50, BRIP1, PALB2などがある。これらもそれぞれが重要な代謝機構を担っている。このように、遺伝子変異によって腫瘍細胞には好都合な機構が作動している上に、悪液質という故障した回路が作動した状態では、修復は極めて困難と思われる。わが国でもがんゲノム情報・先端治療センター(C-CAT)の48,627サンプルを使用して、日本人がん患者におけるドライバー変異の全がん的状況とそれらの臨床的対応の可能性を示した報告が発表された。やはり、TP53の変異頻度は高く、他の多くの遺伝子変異も示された[131]。
8.CAC患者におけるncRNA
- ・筋萎縮、脂肪分解、脂肪褐変および炎症に関係するmiRNAおよびlncRNAは、CAC患者またはCACの動物モデルの筋肉または脂肪組織において調節が解除されている。
- ・調節解除された ncRNA は、がん悪液質患者の循環中または循環エキソソーム中に見られる[132]。
8-Ⅰ)ncRNAの仲間たち、何処に存在する
DNAから転写されたRNAには、タンパク質合成の遺伝情報を含まない多種のnoncoding RNA(ncRNA)が存在する。さらに、筋萎縮、脂肪分解、脂肪褐変および炎症に関係するmiRNAおよびlncRNAは、CAC患者またはCACの動物モデルの筋肉または脂肪組織において調節が解除されている。調節解除された ncRNA は、がん悪液質患者の循環中または循環エキソソーム中に見られ早期がんや諸種疾患の診断や予後判定などに用いられている[133][134]。
miRNA はエキソソームへの封入によって循環移動することもできる[135]。
エキソソームは30~100nmの小さな膜小胞で、タンパク質、RNA、サイトカインおよびその他の分子を効果的に輸送し、循環中の分解を避ける高い安定性を備えている[136][137][138]。
ncRNAには100~200ntを境とした短鎖群のmicro RNA(miRNA)、低分子干渉RNA(siRNA)、核内低分子RNA(snRNA)、piwi相互作用RNA(piRNA)や長鎖群のlong noncoding RNA(lncRNA)、環状RNA(circRNA)などがある。表現型の変動、クロマチン構造、転写、RNA 編集、翻訳などの機能においてその発現の調節を介し、多くの生理学的および病態生理学的プロセスに関与している[139]。
ncRNAの中でもmiRNAの研究は盛んである。miRNAは、血清、唾液、血漿、尿、脳脊髄液などの体液やエキソソーム中に存在し、タンパク質と結合し堅牢な安定性を示す。循環 ncRNAはCACリスクの早期バイオマーカーとしての期待が高い[124][140]。
miRNAは細胞内で生成する20~25ntの短い1本鎖RNAで、数百~数千の塩基の一次転写産物から段階的に切り出され生成する。遺伝子の働きを抑制する機能を持ち、その機能を阻害することでがん細胞の増殖を促進すると考えられている。miRNAは、細胞接触により他の細胞に取り込まれる、または、エキソソームに内包され血液循環により他の細胞へ転送される可能性もあるがんや循環器疾患など多くの疾患の診断・病態生理・治療・予後と広範囲な応用が展開されている。
ヒトゲノムは1900を超えるmiRNAをコードしていると推定されているが[141][142]、現在miRNA遺伝子データベースMirGeneDBに登録されたヒトmiRNAは、567遺伝子、268ファミリーである[143]。
8-Ⅱ)miRNAの塩基多型
miRNAは多種の報告とともに、多くのSNPsやバリアントが存在する。末端1または2塩基が異なる多型もしばしば見出される。この多型の意義は不明であったが、2014年、理化学研究所のグループが、miR-21の多型に伴う分解機構の異常による蓄積を解明し、がん細胞の増殖や浸潤に寄与する機構を解明した[144]。miR-21はPTEN, PDCD4, Bcl-2, RECKなど数多くの腫瘍抑制遺伝子を標的とし、その機能を阻害することでがん細胞の増殖を促進すると考えられている。
miRNAはタンパク質との結合により体液中での安定性が高く、がんの発生機序、増殖、浸潤、転移などに深くかかわる機能を有する。このため、がんに派生する悪液質との強い関連性も推察できる[131][145]。
最近では、ncRNAは、短鎖のmicroRNA類のみではなく、200nt以上の長鎖のlncRNA や circRNAなども同様に、CAC発症に関与する可能性が示唆されている。これは動物モデルやCAC患者でも観察されており、その治療への応用の可能性が期待されている。がんおよびCACにおける全身性炎症の活性化および維持に関与する循環およびエキソソームncRNAに関する報告はここ数年において急激な増加傾向が見られる。このため論文の撤回も散見される。
8-Ⅲ)miRNA表記法
miRNAの名称は以下に示した(1)~(5)の規定に従って順にハイフォン(-)で連結し表記する。これは、新規発見時も同様の扱いである[144]。
Nucleic Acids Res. 2008 Jan;36(Database issue): D154-8.
miRNA:miRNA - Wikipedia(:microRNA - Wikipedia)[146]より引用
- (1)生物の種類を定義する。
- (2)構造のタイプを定義する。
- (3)塩基配列の登録番号を定義する。
- (4)生成過程の由来を定義する。
- (5)miRNAの名称を定義する。
ショウジョウバエ(Drosophila melanogaster)型miRNAの場合、dmeの略称を使用する。マウス(Mus musculus)型miRNAの場合、mmuの略称を使用する。ヒト(Homo sapiens)型miRNAの場合、hsaの略称を使用する。
前駆体の場合、mirを使用する。成熟体の場合、miRを使用する。一部例外も存在する。
同定された順に番号を付ける。類似した配列の場合、番号の後ろに小文字のアルファベットを付ける。
異なる遺伝子座を由来とする場合、数字で区別する。異なる前駆体を由来とする場合、5'末端側の鎖を5p、3'末端側の鎖を3pとして区別する。定義しない場合もある。
(1)~(4)で定義した因子をハイフン“-”でつなげる。例)dme-let-7、mmu-mir-1-1およびhsa-miR-15a-5pなど
8-Ⅳ)がん悪液質におけるmiRNAと動物研究
CACにおける病態解析は患者データの入手が困難なため動物データへの依存が高い。このため、解析結果をヒトに応用するには動物特性、エージング、性差などを考慮した慎重な考察が求められる。また、悪液質の実験も主要な病理像である骨格筋消耗や発症機構への集約感はぬぐえない実情であり、本態の悪液質発症データも待望される。
Leeらは、悪液質を発症したルイス肺がんを有する8週齢C57BL/6Jマウスの前脛骨筋のmiRNA分析により、発現の異なる9つのmiRNA(miR-147-3p, miR-299a-3p, miR-1933-3p, miR-511-3p, miR-3473d, miR-223-3p, miR-431-5p, miR-665-3p, miR -205-3p)を示した[147]。これらは、細胞シグナル伝達や成長、炎症反応などの多くの機能に関与し、異化筋状態の促進の関与が示唆されたが、成熟骨格筋での検証が必要とされる[147]。
Fernandezらは、ルイス肺がんおよび悪液質を患うマウスの前脛骨筋のmRNA および miRNA 発現プロファイルを分析した[148]。細胞外マトリックス、タンパク質分解、および炎症反応に関与すると考えられる遺伝子が同定された。さらに転写因子の結合部位を分析した結果、NF-κB, Stat3, AP-1,FoxOなど、萎縮に関連する転写因子の活性化が示された。 mRNA-miRNA発現プロファイルの統合は、miRNAによる転写後調節を示し、悪液質での筋萎縮における細胞外マトリックスのリモデリングの重要な役割を示唆した[148]。
(miRNAの各論的発現の増減についてはKottorouのレビュー[131]を参照。)
8-Ⅴ)がん性悪液質患者のmiRNA
MyomiRは、筋肉の発達や機能に関与する骨格筋で高い発現レベルを示すmiRNAのセットで具体的には、miR-133a, miR-133b, miR-499, miR-1, miR-206, miR-486, miR-208bが含まれる[149]。今後、研究の進展によりmiRNAは増える可能性はある。
MyomiRは、筋原性調節因子によって転写制御され、骨格筋の成長と筋肉の発達および維持を制御する。miR-1とmiR-206は筋肉の分化を促進し、miR-133は筋肉細胞の増殖を助ける。miR-486は筋肉の再生を促進し、損傷からの回復をはかる。miR-208とmiR-499は心筋の機能を維持する。この様に、MyomiRは筋肉の恒常性と機能を維持するために不可欠な分子である。
Srivastavaらは、「骨格筋疾患におけるバイオマーカーおよび治療標的としてのMyomiRsの新たな役割」の研究で、miR-181a, miR-221, miR-382, miR-434, miR-455の発現が減少し、miR-698とmiR-468の発現が増加することを示した[150]。
miRNAは小さな非コードRNAで、骨格筋の発達と病的状態において役割を果たしている。miRNAは、標的mRNAの3'非翻訳領域に結合することで遺伝子発現を制御する。Narasimhanらは、次世代シークエンシングを用い、悪液質がん患者および非悪液質がん患者の腹直筋生検を分析し、CAC患者でアップレギュレーションした筋肉の代謝、筋形成および炎症に関与する8つのmiRNA、すなわちhsa-let-7d-3p, hsa-miR-423-5p, hsa-miR-345-5p, hsa-miR-1296-5p, hsa-miR-532-5p, hsa-miR-3184-3p, hsa-miR-199a-3pおよび hsa-miR-423-3pを明らかにした[151]。
Freireらは [152]、がん悪液質における新しい潜在的なmiRNA調節遺伝子ネットワークを特定した。miRNA/mRNA相互作用miR-27a/Foxo1, miR-27a/Mef2c, miR-27b/Cxcl12, miR-27b/Mef2c, miR-140/Cxcl12, miR-199a/Cav1およびmiR-199a/Junbを示した。これらはCACにおける筋肉の消耗に関与している可能性がある。さらに、この結果から、miR-145aが悪液質筋消耗中の新たな潜在的なFoxO1調節因子であると結論付けた。
さらに、Fernandezらは、miR-29b-3pはCAC誘発性の骨格筋萎縮のバイオマーカーの可能性を報告した[148]。
Kottorouらは、let-7d-3pが筋細胞の増殖と筋原性分化に影響を与えるトランスフェリン受容体経路のダウンレギュレーションを促進する一方、miR-345-5pがNOVおよびCOL1A1をダウンレギュレーションし、その結果CYR61をアップレギュレーションすることを実証した[132]。NOVとCYR61は、インスリン様成長因子1、Akt、およびmTOR 経路に関与しており、タンパク質合成能力を低下させる。
Kottorouらは、同じ研究にて、miR-423-5pとmiR-3184-3pが脂質生合成に関与するSQLEとFADS2をダウンレギュレーションする一方、miR-423-5pはエネルギーバランスとエネルギーバランスに関与するレプチンやその他の遺伝子も制御できることが示された。筋肥大に関与するDLK1をダウンレギュレーションした。さらに、miR-423-3pがカルシウムシグナル伝達障害を促進し、CAMK2Aに影響を与えること、miR-3184-3pがWnt/β-カテニンシグナル伝達に関与し、筋原性分化を減弱させること、およびmiR-3184-3pがBMPR1BおよびGREM1を調節してTGF-3に影響を与えることを示した。βおよびBMPシグナル伝達、さらにmiR-532-5pは、体節の発達に影響を与えるBMPシグナル伝達に関連するSULF1, CNTF(毛様体神経栄養因子)活性に寄与するRPS6KA6、および食欲調節に関与するNPY1Rを標的とすることが実証された。全体的にNarasimhanらは、悪液質がん患者の筋萎縮に関与する経路の調節において、さまざまなmiRNAが重要な役割を果たすことを示唆した[132]。
CACにおける筋肉消耗のタンパク質-タンパク質相互作用ネットワークでは、相互作用成分のほとんどが転写因子およびアポトーシス過程、代謝および炎症反応に関連するタンパク質であることが強調された。さらに、この研究では、がん性悪液質中の筋肉減少に寄与する可能性があるmiR-27a/Foxo1, miR-27a/Mef2c, miR-27b/Cxcl12, miR-27b/Mef2c, miR-140/Cxcl12, miR-199a/Cav1, miR-199a/Junbなどの新しいmiRNA-mRNA相互作用が予測された[152][153]。
Okugawaらは、大腰筋指数が低い大腸がん患者は血清中のmiR-21値が高く、miR-21は臨床医がこれらの患者の栄養戦略を決定する上で有用なパラメータになることを示した[154]。
8-Ⅵ)CAC患者における循環miRNA
CAC患者の循環 miRNA
miRNAは細胞内に局在するだけでなく、細胞から能動的に分泌されたり、さまざまな刺激に反応して細胞膜から放出されたりして、血液やその他の体液中を循環する[155]。ミオスタチンやアルギニンなど、筋肉量と関連付けられている血液バイオマーカーは非常に少ないため、筋肉量減少のモニターに有益である[156][157][158]。循環miRNAは、筋肉量減少だけではなく、筋肉再生、治療の有効性、悪液質の早期検出にも潜在的なバイオマーカーとしても有用である。頭頸部がんでmiR-130aの血漿レベルが低い患者は、TNF-αの血漿レベルが高く、悪液質のリスクが高いことが報告されている[159]。
有用なマイクロRNAの報告は多く、特定には若干の調査を要すると思われる。現段階でまとめられたいくつかのリストの1例として、図9にKottorouら(参考文献[132])の図2「筋肉または脂肪組織、あるいはがん性悪液質を患う患者または動物モデルの循環における調節解除されたncRNA」を和訳引用した。
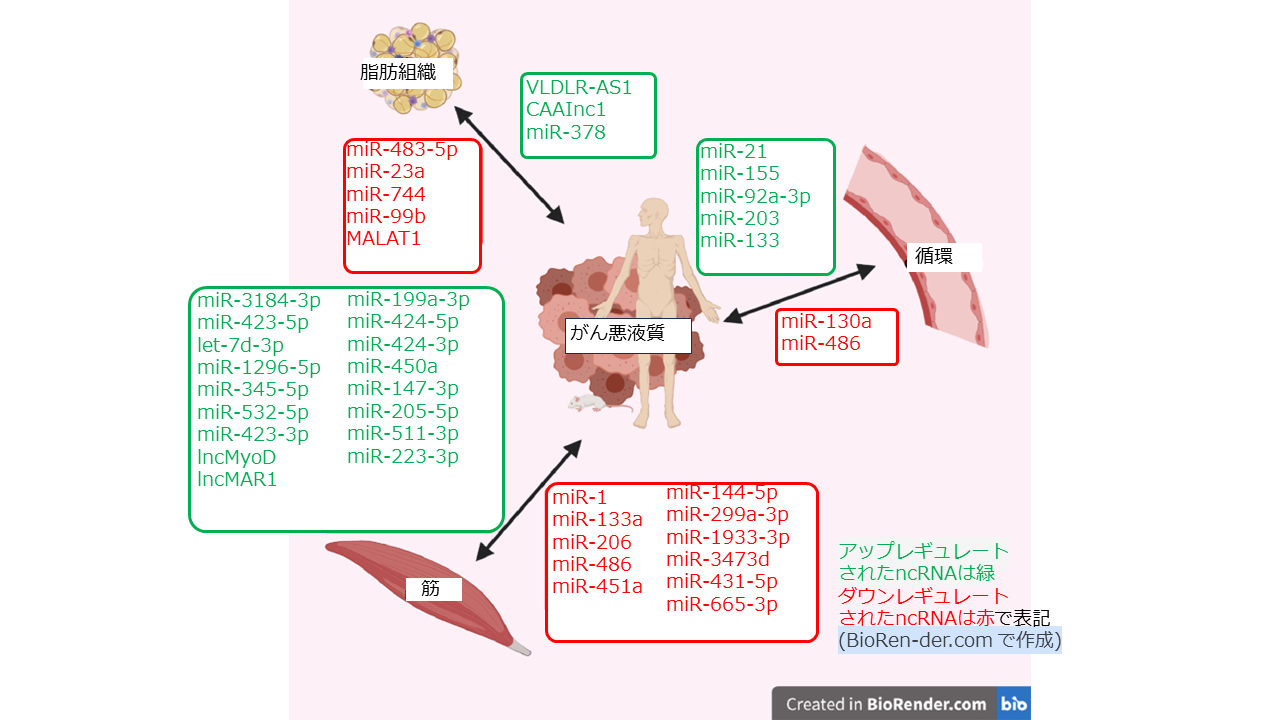
図9 筋肉または脂肪組織、あるいは癌性悪液質を患う患者または
動物モデルの循環における調節解除されたncRNA
Transl Oncol. 2021 Jul;14(7):101101. から和訳引用し一部改変
マイクロRNAは有用性が評価される一方、分析対象試料が組織生検、循環miRNA、エキソソーム中miRNAなどの生体試料種によって分析値は異なるため堅実な認識が求められる。
また、1種類のmiRNAは、多種類のmRNAの遺伝子発現調節に関わっているため、1種類のmRNAの遺伝子発現は、多種類のmiRNAによって調節されていることも認識しておく必要がある。
9.CACを引き起こす原因の追究
CACを引き起こす原因の追究は、万人が望む究極の頂点であり、未だ到達できていないが、いくつかの推論や仮説は生まれている。その一つは[21]に記されたFerraraらの悪液質進行の仮説的時間モデルとPark MAらの[1]に記された、(1)肝臓中心と(2)腫瘍中心の仮説がある。
9-Ⅰ)悪液質進行の仮説的時間モデル
Ferraraらは、悪液質の進行に伴う臓器/組織の混乱のタイムラインの重要性を説き、参考文献[21]に、文献上からの報告データを基に「前臨床モデルと患者からの所見を統合した悪液質進行の仮説的時間モデル」を図示した。
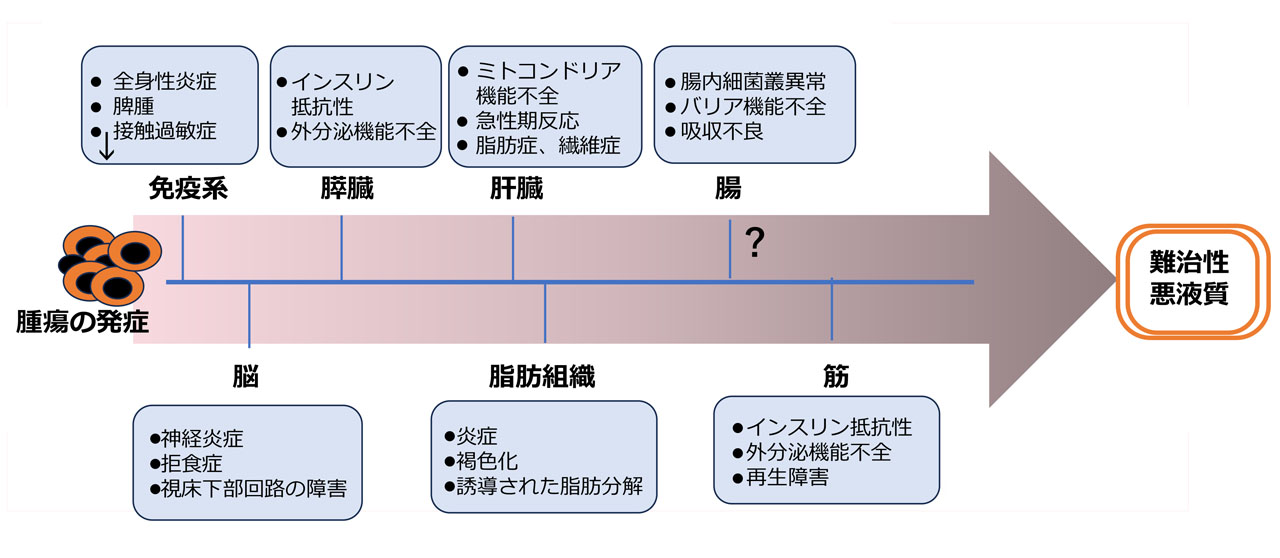
図10 前臨床モデルと患者からの所見を統合した悪液質進行の仮説的時間モデル
Front Cell Dev Biol. 2022 Sep 8;10:960341.より和訳引用し改変
以下は、図の説明文を和訳引用し改変。
- 『文献で報告されたデータに基づいて、悪液質の進行に伴う臓器/組織の混乱のタイムラインを提案し、悪液質の主要な病理学的プロセスの一つと考えられている骨格筋の消耗は後期の出来事であるように見えることを強調したい。』
- 『1.腫瘍の増殖に応じて、最初に影響を受ける組織は免疫系であり、悪液質プロセスおよび全身性炎症の確立における中心となる。
2.食欲不振は悪液質進行の初期事象であることがいくつかの報告で示されており、食欲不振の原因は未だ解明されていないものの、炎症性メディエーターと腫瘍由来の因子が食欲不振に重要な役割を果たしているようである。
3.腫瘍発生に対する宿主の適応におけるもう 1つの初期イベントは膵臓の内分泌機能(インスリン抵抗性)と外分泌機能の両方の障害であり、腫瘍の増殖と悪液質の発生の両方に影響を与える。したがって、膵臓酵素の変化は脂肪組織の消耗に直接関連しており、それが、がん関連悪液質の初期事象である可能性があることを示している。いくつかの研究ではミトコンドリア機能不全や急性期反応などの肝臓の変化も、腫瘍発生後の早期に介入する一方、脂肪変性や線維化はその後に現れる可能性があることを示している。
4.最も研究されている悪液質の組織である脂肪組織と骨格筋は、骨格筋の消耗に先立って脂肪組織の変化が起こり、緊密なクロストークに関与している。炎症誘発性サイトカインなどのさまざまな刺激によって引き起こされる脂肪分解は、循環中の遊離脂肪酸の放出を誘発する。
5.骨格筋における遊離脂肪酸の過剰な酸化は、筋肉の消耗を促す最も初期の現象の一つと考えられ、脂肪分解と筋肉の分解との機能的および時間的な関連性を裏付けている。
6.最後に、腸の恒常性の変化が悪液質の発症に深く寄与していることが報告されているが、悪液質の発症時に腸の変化が現れる可能性を示唆する証拠はあるにもかかわらず、悪液質症候群のどの段階で腸の機能不全が発生するかは不明である。』と述べている。
9-Ⅱ)がん性悪液質を引き起こす原因
【仮説:肝臓中心、腫瘍中心】
(参考文献[1]から和訳引用)
『がん性悪液質を引き起こす原因について、(1)肝臓中心、(2)腫瘍中心という2つの仮説を捉えることに重点を置く。肝臓中心のモデルでは、患者の代謝要求と腫瘍量の合計が、肝臓が血液に栄養素を補給し、代謝物や毒素を除去して処理する能力を超えたときに、CAC が進行すると考えられている [160][161] [162] [163] [164] [165]。この過剰需要を満たすために、肝臓は筋肉と脂肪組織の分解を指示して、新たな供給を誘発する。がんがない場合、これは肺、皮膚、または心臓の損傷に対する、急性の過剰需要に対する身体の適応反応である可能性がある。しかし、腫瘍の成長が続くと、肝臓への過剰需要が時間とともに増加し、筋肉と脂肪組織の進行性の喪失を引き起こす。対照的に、腫瘍中心モデルでは、悪液質は炎症誘発性サイトカインと、抗炎症性サイトカインの不均衡の結果として生じるとみられている[8][164][166][167]。
悪液質は肝臓中心モデルでは、資源補充の不均衡として定義される[24] [168]。必要な補充の一定速度を超えると、肝機能は速度制限となり、筋肉を消耗し始める。肝臓中心モデルでは、腫瘍による資源の需要と潜在的に有毒な代謝物の放出に起因する代謝調節異常が、肝臓の重要な機能に直接過負荷をかけると仮定している。その結果、肝臓は最初に筋肉に、次に脂肪組織にアミノ酸、そして脂肪酸の信号を送り、この肝機能への過負荷がCACに見られる消耗を直接引き起こす。あるいは、この同じ現象論的モデルに腫瘍中心仮説を組み込むことも可能であり、腫瘍がサイトカインストームを介して直接CACを誘発し、全身性炎症を引き起こし[169]、その結果、筋肉と脂肪組織の恒常性が調節不全となり、慢性的な消耗が生じる[24]。これは腫瘍によるドローダウン率の増加として現れ、誘発された全身性炎症の関数として悪液質率を増加させる。』
おわりに
「がん」といえば死の宣告と同義に捉えられた時期もあったが、近年では、がん治療法も手術療法、化学療法(抗がん剤)、放射線療法、光免疫療法、免疫療法、ウイルス療法、がんワクチン、温熱療法などなど、選択肢の広がりとともに治療成績は急激にあがり、高い5年生存率の向上が得られ、同時に寛解例も散見されるようになった。しかし、これも発症初期の検出例は治療成果を生みやすいが、再発、転移、末期がんでは治療困難な疾患群に属する。さらにこれに悪液質を併発した場合、予後不良となりかねない。近年では、診断学分野においても血液、尿、唾液、便、脳脊髄液などのliquid biopsyによる、がんの超早期検出や遺伝情報による適正治療の選択などが実施され多大な成果を挙げている。中でもマイクロRNAを含むncRNAグループの成果は目覚ましく、未知な機能満載の感を抱く。悪液質のより早期の臨床的介入可能なバイオマーカーを探索し、がん悪液質に対する多様な組織と代謝経路を解明し、集学的治療のさらなる研究を深めると同時に臨床検査にがん悪液質の早期診断項目が導入され、治療体系が確立したら、「がん」を患っても、今日の治療法の進展と相まって「がん」を恐れることなく治療に専念し、多くの患者が寛解できる日もそう遠い夢ではないかもしれない。

〔執筆に関連した文献や資料の翻訳には「Google翻訳」を主体に、一部「Deep L」を用いた。また、語彙の解釈には「Copilot」を活用した。さらに、校正の労を頂いた(株)エイアンドティースタッフの方に重ねて深謝致します。〕
参考文献・資料
- Park MA, Whelan CJ, et al., Cancers (Basel). 2024 Jun 27;16(13):2364.
- Anker MS, Holcomb R, et al., J Cachexia Sarcopenia Muscle. 2019 Feb;10(1):22-34.
- Tan BH, Fearon KC. Curr Opin Clin Nutr Metab Care. 2008 Jul;11(4):400-7.
- Jafri S.H.R., Previgliano C., et al., Clin. Med. Insights Oncol. 2015;9:S30891.
- Freire PP, Fernandez GJ, et al. J Cachexia Sarcopenia Muscle [Internet] 2020; 11:947–961.
- Fearon K, Arends J. et al. Nat. Rev. Clin. Oncol. 2013:90–99.
- Meza-Valderrama D, Marco E. et al., Nutrients 2021, 13, 761.
- Nishikawa H, Goto M. et al., Int. J. Mol. Sci. 2021, 22, 8491.
- Chasen M.R., Bhargava R., Support. Care Cancer. 2009; 17:1345–1351.
- Argilés, J.M., Stemmler, B., et al. Nat Rev Endocrinol 15, 9–20 (2019).
- Ferrara M, Samaden M, et al., Front Cell Dev Biol. 2022 Sep 8;10:960341.
- Mariean CR, Tiucă OM, et al. Cancers (Basel). 2023 Nov 26;15(23):5590.
- Hariyanto TI., Kurniawan A. Cachexia in Cancer Patients: Systematic Literature Review. Asian J. Oncol. 2020; 06:107–115. doi: 10.1055/s-0040-1713701.
- Evans, W.J., Morley J.E., et al. A New Definition. Clin. Nutr. 2008, 27, 793–799.
- Fearon K, Strasser F, et al., Lancet Oncol. 2011 May;12(5):489-95.
- Cederholm, T., Compher C et al., Clin Nutr. 2019 Jun;38(3):1480-1481.
- Rier, H.N., Jager A., et al., Oncologist. 2016 Nov; 21(11): 1396–1409.
- Han, J., Harrison L., et al. EJNMMI Res. 2021, 11, 94.
- Panebianco, C., Villani A., et al., Int. J. Mol. Sci. 2023, 24, 1849.
- Kottorou A., Dimitrakopoulos FI., et al., Transl Oncol. 2021 Jul;14(7):101101.
- Berriel Diaz, M., Rohm, M. et al., Nat Metab 6, 2222–2245 (2024).
- Bannangkoon, K., Hongsakul K., et al. Sci Rep 13, 3978 (2023).
- Lortie J, Rush B., et al., Front Rehabil Sci. 2022 May 30;3:896114.
- Baracos V. E., Martin L., et al., (2018). Nat. Rev. Dis. Prim. 4, 17105. 10.1038/ nrdp. 2017.105
- Laird B., Fallon M. (2017). Ann. Oncol. 28, 2054–2055. 10.1093/annonc/mdx349
- Wu H., Ballantyne C. M. (2017). J. Clin. Invest. 127, 43–54. 10.1172/JCI88880
- Cortiula F., Hendriks LEL., et al., J Cachexia Sarcopenia Muscle. 2022 Feb;13(1):55-67.
- Tracey KJ, Lowry SF, et al., (1988). J. Infect. Dis. 157, 413–420.
- Graziano F, Ruzzo A, et al., (2005). J. Clin. Oncol. 23, 2339–2345.
- Zhang D, Zheng H, et al., BMC Cancer. 2007 Mar 14; 7:45.
- Scheede-Bergdahl C, Watt HL, et al. (2012). Clin. Nutr. 31, 85–88.
- Narsale AA, Carson JA, (2014). Curr. Opin. Support. Palliat. Care 8, 321–327.
- Pettersen K, Andersen S, et al. (2017). Sci. Rep. 7, 2046. 10.1038/s41598-017-02088-2
- Baltgalvis KA, Berger FG, et al., (2008). Am. J. Physiol. Regul. Integr. Comp. Physiol. 294, R393–R401
- Agca S, Kir S. FEBS J. 2024 Sep;291(18):4009-4023.
- Zimmers TA, Fishel ML, et al., (2016). Semin. Cell Dev. Biol. 54, 28–41.
- Lavine KJ, Sierra OL, Heart Fail Rev. 2017 Mar;22(2):179-189.
- White JP. Transl Cancer Res. 2017 Mar;6(Suppl 2): S280-S285.
- Jones A, Friedrich K, et al, EMBO Mol Med. 2013 Feb;5(2):294-308.
- Gonçalves DC, Lira FS, et al. (2019). Clin. Nutr. 38, 2219–2230.
- Mahon OR, Dunne A, (2018). Front. Immunol. 9, 1145. 10.3389/fimmu.2018.01145
- Costa RGF, Caro PL, et al. (2019). J. Cachexia Sarcopenia Muscle 10, 1116–1127.
- Smith MA, Moylan JS, et al., (2007). Am. J. Physiol. Cell Physiol. 293, C1947–C1952.
- Kumar A, Bhatnagar S, et al., (2012). Curr. Opin. Clin. Nutr. Metab. Care 15, 233–239.
- Johnston AJ, Murphy KT, et al. (2015). Cell 162, 1365–1378.
- Kandarian SC, Nosacka RL, et al. (2018). J. Cachexia Sarcopenia Muscle 9, 1109–1120.
- Hishida A, Okugawa Y, et al. (2019). Oncol. Lett. 17, 623–629.
- Petruzzelli M, Ferrer M, et al. (2022). Cancers 14, 963.
- Erdem M, Möckel D, et al. (2019). J. Cachexia Sarcopenia Muscle 10, 1128–1142.
- Cuenca AG, Cuenca AL, et al. (2014). J. Immunol. 192, 6111–6119.
- Narsale AA., Moya R, et al. (2019). J. Cachexia Sarcopenia Muscle 10, 827–843.
- Faber J, Vos AP, et al. (2009). Oncol. Rep. 22, 1403–1406.
- Ju, J.E., Kim, MS., et al. Potential role of immunological factors in early diagnosis of cancer cachexia in C26 tumor-bearing mice. Appl Biol Chem 62, 3 (2019).
https://doi.org/10.1186/s13765-019-0417-5 Google - Argilés, J, Busquets S, et al. Nat Rev Cancer 14, 754–762 (2014).
- Setiawan T, Sari IN, et al. J Hematol Oncol 16, 54 (2023).
- Relaix F, Bencze M, et al. Nat Commun. 2021 Jan 29;12(1):692.
- Blazev R, Ashwood C, et al. Mol Cell Proteomics. 2021;20: 100030.
- Jorgenson KW, Phillips SM, et al. Cells. 2020;9(7):1658.
- Coudert L, Osseni A, et al. BMC Biol. 2021;19(1):153.
- McKinnell IW, Rudnicki MA. Cell. 2004;119(7):907–910.
- Homa-Mlak I, Pigoń-Zając D, et al., J Pers Med. 2022 Aug 31;12(9):1438.
- Pin F, Minero VG, et al. Front Physiol. 2017;8:213.
- Bonetto A, Aydogdu T, et al. Am J Physiol Endocrinol Metab. 2012;303(3):E410–E421.
- Braun TP, Zhu X, et al. J Exp Med. 2011;208(12):2449–2463.
- Li YP, Chen Y ,et al., FASEB J. 2005 Mar;19(3):362-70.
- Camperi A, Pin F, et al. Oncotarget. 2017;8(13):21778–21793.
- Judge SM, Wu CL, et al. BMC Cancer. 2014; 14:997.
- Marzetti E, Calvani R, et al., Aging Clin Exp Res. 2017 Feb;29(1):35-42.
- Baracos VE, Mazurak VC, et al., Ann Palliat Med. 2019 Jan;8(1):3-12.
- Schmitt TL, Martignoni ME, et al. J Mol Med (Berl) 2007;85(6):647–654.
- Silva KA, Dong J, et al. J Biol Chem. 2015;290(17):11177–11187.
- Sugiyama M, Yamaki A, et al., Regul Pept. 2012 Oct 10;178(1-3):21-8.
- Yang J, Zhang Z, et al. Gastroenterology. 2019;156(3):722–734.
- Chen JL, Walton KL, et al., FASEB J. 2014 Apr;28(4):1711-23.
- Glass DJ. Int J Biochem Cell Biol. 2005;37(10):1974–1984.
- Zhou X, Wang JL, et al., Cell. 2010 Aug 20;142(4):531-43.
- Benny Klimek ME, Aydogdu T, et al. Biochem Biophys Res Commun. 2010;391(3):1548–1554.
- Loumaye A, de Barsy M, et al. J Clin Endocrinol Metab. 2015;100(5):2030–2038.
- McPherron AC, Lawler AM, Lee SJ. Nature. 1997;387(6628):83–90.
- Lee SJ, McPherron AC. Proc Natl Acad Sci U S A. 2001;98(16):9306–9311.
- Gallot YS, Durieux AC, et al. Cancer Res. 2014;74(24):7344–7356.
- Goodman CA, McNally RM, et al. Mol Endocrinol. 2013;27(11):1946–1957.
- Thoma A, Lightfoot AP. Adv Exp Med Biol. 2018;1088:267–279.
- Li YP, Schwartz RJ, et al., FASEB J. 1998 Jul;12(10):871-80.
- Hunter RB, Stevenson E, et al., FASEB J. 2002 Apr;16(6):529-38.
- Di Marco S, Mazroui R, et al. Mol Cell Biol. 2005;25(15):6533–6545.
- Guttridge DC, Mayo MW, et al. Science. 2000;289(5488):2363–2366.
- Fry CS, Nayeem SZ, et al. Physiol Rep, 2016. 4(21).
- Ma JF, Sanchez BJ, EMBO Mol Med. 2017 May;9(5):622-637.
- White JP, Puppa MJ, et al. Am J Physiol Endocrinol Metab. 2013;304(10): E1042–E1052.
- Powrozek T, Mlak R, et al. J Cancer Res Clin Oncol. 2018;144(8):1423–1434.
- Siddiqui RA, Williams JF. Biochem Int. 1990;20(4):787–797.
- Matsuyama T, Ishikawa T, et al. Int J Cancer. 2015;137(11):2558–2565.
- Kami K, Senba E. J Histochem Cytochem. 2002;50(12):1579–1589.
- Cramer Z, Sadek J, et al. Sci Rep. 2018;8(1):8414.
- Sala D, Sacco A. Curr Opin Clin Nutr Metab Care. 2016;19(3):171–176.
- Eskiler GG, Bezdegumeli E, et al. Bratisl Lek Listy. 2019;66(11):819–826.
- Bonetto A, Aydogdu T, et al. PLoS ONE. 2011;6(7): e22538.
- Ying L, Yao Y, et al., Am J Physiol Cell Physiol. 2022 May 1;322(5):C814-C824.
- Pin F, Barreto R, et al. J Cachexia Sarcopenia Muscle. 2018;9(4):685–700.
- Hsu MY, Porporato PE, Wyart E. Methods Mol Biol. 2019;1928:337–352.
- Fontes-Oliveira CC, Busquets S, et al. Biochim Biophys Acta. 2013;1830(3):2770–2778.
- Julienne CM, Dumas JF, et al. J Cachexia Sarcopenia Muscle. 2012;3(4):265–275.
- Romanello V, Guadagnin E, et al., EMBO J. 2010 May 19;29(10):1774-85.
- Cao Z, Andrew M, et al. JCSM Rapid Commun. 2021 4, 166–186.
https://doi.org/10.1002/rco2.30 - Nogueira-Ferreira R, Sousa-Nunes F, et al., Inflamm Res. 2022 Aug;71(7-8):771-783.
- Laird BJ, Kaasa S, et al., Clin Cancer Res. 2013 Oct 1;19(19):5456-64.
- Tong T., Guan Y, et al., Front. Oncol. 2020; 10:1541.
- Barker T, Fulde G, et al. Sci Rep 10, 7535 (2020).
- Cao Z, Zhao K, et al., Int J Mol Sci. 2021 Apr 26;22(9):4501.
- Ebadi, M., Mazurak, V. C. (2015). Mediat. Inflamm., 820934.
- Loumaye A, de Barsy M, et al. (2017). J. Cachexia Sarcopenia Muscle 8, 768–777.
- Cole CL, Kleckner IR, et al., JCSM Clin Rep. 2018 Jul-Dec;3(2):e00065.
- Ahmed DS, Isnard S, et al., J Cancer. 2021 Jan 1;12(4):1125-1132.
- Kir, S., White JP, et al. Nature. 2014 Sep 4;513(7516):100-4.
- Saito H, Inagaki Y, et al., Cancer Sci. 2007 Oct;98(10):1563-9.
- Olson, B., Zhu X, et al. Nat Commun 12, 2057 (2021).
- Olson B, Diba P, et al., Cancers (Basel). 2021 Aug 7;13(16):3990.
- Yeom E, Shin H, et al., Nat Cell Biol. 2021 Feb;23(2):172-183.
- Sanders PM, Tisdale MJ. Br J Cancer. 2004 Mar 22;90(6):1274-8.
- Morigny P, Zuber J, et al., J Cachexia Sarcopenia Muscle. 2020 Dec;11(6):1459-1475.
- O’Connell, T. M., Golzarri-Arroyo, L., et al. (2021). Front. Cell Dev. Biol. 9, 720096.
- Yeom, E., Yu, K. Exp Mol Med 54, 426–432 (2022).
- Santos, J. M. O., Peixoto da Silva, et al. (2020). Cancers 12, E1004.
- Miller, J.; et al., Cancers 2019, 11, 1594.
- Cala MP, Agulló-Ortuño MT, et al., J Cachexia Sarcopenia Muscle. 2018 Apr;9(2):348-357.
- Cui P, Li X, et al., (2022). Front. Mol. Biosci. 9, 789889.
- Lerner L, Tao J, et al., J Cachexia Sarcopenia Muscle. 2016 Sep;7(4):467-82.
- Suriben R, Chen M, et al., Nat Med. 2020 Aug;26(8):1264-1270.
- López-Otín C, Kroemer G. Hallmarks of Health. Cell. 2021 Jan 7;184(1):33-63.
- Horie S, Saito Y, et al., Cancer Discov. 2024 May 1;14(5):786-803.
- Ru Q, Chen L, et al., J Transl Med. 2024 Apr 30;22(1):408.
- Matsuzaki J, Kato K, et al., JNCI Cancer Spectr. 2023 Jan 3;7(1): pkac080.
- Kim T, Croce CM. Exp Mol Med. 2023 Jul;55(7):1314-1321.
- Nie M, Deng ZL, et al., Biomed Res Int. 2015;2015:676575.
- Wang H, Wang B., Biomed Rep. 2016 Sep;5(3):296-300.
- H Rashed M, Bayraktar E, et al., Int J Mol Sci. 2017 Mar 2;18(3):538.
- Iraci N, Leonardi T, et al., Int J Mol Sci. 2016 Feb 6;17(2):171.
- John S. Mattick, Igor V. Makunin, Human Molecular Genetics, Volume 15, Issue suppl_1, 15 April 2006, Pages R17–R29,
https://doi.org/10.1093/hmg/ddl046 - Donzelli S, Farneti A, et al., Front Cell Dev Biol. 2020 Apr 21;8:257.
- Homo sapiens miRNAs in the miRBase at Manchester University
- Alles J, Fehlmann T, et al. Nucleic Acids Research. 47 (7): 3353–3364.
- Fromm B, Domanska D, et al. Nucleic Acids Res. 2020 Jan 8;48(D1): D132-D141.
- Boele J, Persson H, et al., Proc Natl Acad Sci U S A. 2014 Aug 5;111(31):11467-72.
- Zhou X, Hu S, et al., Precis Clin Med. 2021 Apr 23;4(2):136-147.
- miRNA - Wikipedia(:microRNA - Wikipedia)
- Lee DE, Brown JL, et al., Physiol Genomics. 2017 May 1;49(5):253-260.
- Fernandez GJ, Ferreira JH, et al., Front Genet. 2020 May 29; 11:541.
- Giagnorio E, Malacarne C, et al. J Cell Sci. 2021 Jun 15;134(12): jcs258349.
- Srivastava S, Rathor R, et al. Am J Physiol Cell Physiol. 2021 Nov 1;321(5):C859-C875.
- Narasimhan A, Ghosh S, et al. J Cachexia Sarcopenia Muscle 8: 405–416, 2017.
- Freire PP, Fernandez GJ, et al., Int J Mol Sci. 2019 Apr 22;20(8):1962.
- Narasimhan A, Shahda S, et al., Cancers (Basel). 2020 Dec 15;12(12):3787.
- Okugawa Y, Yao L, et al., Oncol Rep. 2018 Apr;39(4):1555-1564.
- Gayosso-Gómez LV, Ortiz-Quintero B. Diagnostics (Basel). 2021 Mar 2;11(3):421.
- Scherbakov N, Knops M, et al., J Cachexia Sarcopenia Muscle. 2016 Mar;7(1):60-7.
- Nishikawa H, Enomoto H, et al., J Cachexia Sarcopenia Muscle. 2017 Dec;8(6):915-925.
- Calvani R, Marini F, et al., J Cachexia Sarcopenia Muscle. 2015 Dec;6(4):278-86.
- Powrózek T, Mlak R, et al., Cancers (Basel). 2018 Aug 30;10(9):294.
- Argilés JM, Moore-Carrasco R, et al., Drug Discov Today. 2003 Sep 15;8(18):838-44.
- Munro HN. J Am Diet Assoc. 1977 Oct;71(4):380-4. PMID: 908798.
- Bossi P, Delrio P, Nutrients. 2021 Jun 9;13(6):1980.
- Poulia KA, Sarantis P, et al., Nutrients. 2020 May 26;12(6):1543.
- Siddiqui JA, Pothuraju R, et al., Cytokine Growth Factor Rev. 2022 Apr;64:71-83.
- Hall KD, Baracos VE. Curr Opin Clin Nutr Metab Care. 2008 May;11(3):214-21.
- Talbert EE, Guttridge DC. Trends Cancer. 2022 May;8(5):397-403.
- Rupert JE, Narasimhan A, et al., J Exp Med. 2021 Jun 7;218(6): e20190450.
- Rohm M, Zeigerer A, et al., EMBO Rep. 2019 Apr;20(4): e47258.
- Macciò A, Madeddu C, et al., Br J Cancer. 2023 May;128(9):1609-1610.
イラスト/菅原 智美
2025.03